上海艾跃(Active Motif)生物科技有限公司品牌商
9 年
手机商铺
- NaN
- 0.3999999999999999
- 0.3999999999999999
- 2.4
- 2.4
上海艾跃(Active Motif)生物科技有限公司
入驻年限:9 年
- 联系人:
Active Motif
- 所在地区:
上海 闵行区
- 业务范围:
试剂、实验室仪器 / 设备、技术服务、抗体
- 经营模式:
生产厂商 经销商
推荐产品
公司新闻/正文
如何从少量的临床样品中获取大量的组蛋白修饰数据?质谱法!
1854 人阅读发布时间:2022-11-29 15:24
量化临床样本的组蛋白修饰水平有助于研究新的疾病特异性表观遗传机制,创建患者特异性治疗策略,并能够将患者准确分为不同的治疗组。虽说现有的细胞系为研究疾病组蛋白修饰提供了丰富的细胞材料,但要深入准确地研究疾病样本的组蛋白修饰,绕不开临床样本的使用。
在临床样本中提取、分离和定量蛋白质的过程中每一步都面临着挑战,临床样本体积小,并且很多组织区域需人工手动操作,使得研究临床样本时,需要更加有效和精确的实验技术。
在癌症研究中,从少量样本中获得大量组蛋白修饰数据将有助于分析早期癌症病变、微转移、肿瘤内异质性、肿瘤治疗反应以及肿瘤细胞亚群之间的相互作用。
质谱法——临床样本解决方案的“一小步”
来自IRCCS的Roberta Noberini和Tiziana Bonaldi实验室的研究人员在前期的工作中,开发出用于临床FFPE组织样本、冰冻组织、和原代细胞中分析组蛋白修饰的组蛋白富集方法(Colzani等人,2014,Noberini等人,2017,Noberni等人,2020,和Restellini等人,2019)。
值得注意的是,他们的方法采用了质谱法,通过测量样品中分子的质荷比,用来识别样品的成分。但质谱法需要单独的实验方法对组蛋白H3(在凝胶中消化,Colzani等人,2014)和组蛋白H4(在溶液中消化,Restellini等人,2018)进行处理,以最大限度地覆盖组蛋白修饰。使用这种方法可监测数十种组蛋白H3和H4修饰,因此研究人员能够观察到全面的组蛋白修饰变化,并可能发现预期之外的组蛋白修饰信息。
临床样本的质谱分析
质谱法在临床方面的应用已在肿瘤和正常组织(Noberini 等人, 2019),以及各种乳腺亚型(Fowler等人, 2011)中得到验证,能够从这些样本中获得对应的表观遗传模式。然而,为了获得这些信息,往往需要大量的起始样本,这也成为相关研究的一大限制因素。
目前,手动/激光显微解剖技术可以有效的减少样本的需求量(可降低至500,000 cells),但依旧对临床样本来说是一个巨大的挑战,为了解决这一问题,Noberini和同事共同开发出了新的实验方法,该方法基于激光显微切割的组织区域将样本需求量降到了更低(大约1000个细胞)。

第一步——“凝胶内PRO-PIC”消化方案
作者开发了一种高效的凝胶内消化方案(PRO-PIC)用于处理临床样本,使得质谱能够检测起始量稀少的临床样本中的组蛋白修饰。与其他相关技术相比,该方法消除了溶液内消化造成的质谱污染,减少了使用的组织量,支持更多数量的修饰肽的定量,并增加了低丰度组蛋白修饰的检测。总的来说,凝胶内PRO-PIC方案允许检测具有挑战性的组蛋白修饰(例如,H3K4me2/me3或修饰的H4K20),有效区分常见乙酰化的赖氨酸残基(例如H3K27ac、H3K36ac和H3K37ac)。
此方法成功的从三阴性乳腺癌细胞核提取物中分离的5 ng肽中检测到多达38个组蛋白修饰。此外,用组蛋白去乙酰化酶抑制剂治疗三阴性乳腺癌细胞后诱导的组蛋白修饰改变可以通过质谱容易且可重复地展示出乙酰化组蛋白衍生肽的普遍增加。
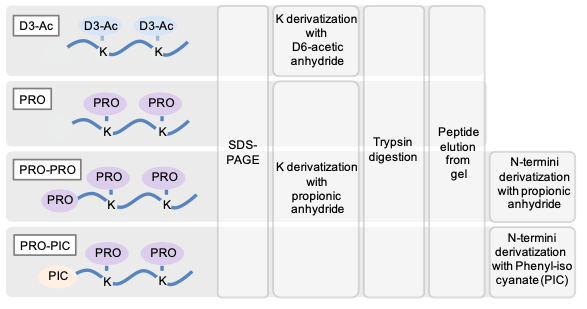
用于凝胶消化组蛋白的方案示意图
在后续的评估步骤中,凝胶内PRO-PIC被用于手动显微切割(500,00和100,000个细胞)、激光显微切割(20,000、5,000、2,500和1,000个细胞)和FFPE小鼠胰腺组织切片,随后研究者利用质谱分析组蛋白修饰。从结果上来看,此方法能够检测到大量和少量细胞样品中的大多数组蛋白修饰。此外,作者还注意到,在所有条件下,肽段比例都具有极高的相关性。这些结果表明,使用PRO-PIC消化的质谱工作流程可以很容易地应用于非常小的组织样品,从而能够从难以获得、稀少、珍贵的样本中收集准确的数据信息。
在人类乳腺癌样本中的验证
最后的验证实验涉及将凝胶内PRO-PIC方案应用于Luminal A亚型乳腺癌患者分离的冷冻激光显微解剖组织,包括各种肿瘤和相邻的正常组织区域。总体而言,他们观察到H3K14ac、单乙酰化H4和H4K20me3的丢失,以及所有肿瘤区域乙酰化H3肽的减少,值得注意的是,H3K9/K14和H4尾部乙酰化水平显示出异质水平。四乙酰化组蛋白H4尾部肽的水平在Luminal A肿瘤组织中显示出增加,然而,该肽的水平在不同的肿瘤样品间显示出差异。此外,作者未能在三阴性乳腺癌、卵巢、头颈部肿瘤中检测到组蛋白修饰的这种改变模式,这表明四乙酰化组蛋白H4尾部肽水平的增加似乎具有肿瘤亚型特异性,并在同一肿瘤的不同区域是可区分的。
作者后续通过取样包含肿瘤细胞、肿瘤浸润淋巴细胞和肿瘤区域外淋巴细胞的区域,评估了三阴性乳腺癌患者样本中的组织异质性。肿瘤细胞显示H4K20me3水平降低,未修饰的H3和H4肽增加,在比较肿瘤细胞和正常细胞时也发现了这种模式,这可能是由肿瘤细胞的高度增殖性所引发的。与淋巴细胞相比,肿瘤细胞显示出四乙酰化组蛋白H4尾肽水平增加,但除了四乙酰化组蛋白H4尾肽外,两种不同的淋巴细胞类型在组蛋白修饰水平上保持相似。
结论
总的来说,这项令人着迷的研究表明,质谱法从小规模的临床样本中获得大量组蛋白修饰数据的技术可行性。首次描述了该技术在肿瘤亚群中检测组蛋白修饰水平的能力,这可能会使该领域在我们对肿瘤转移、肿瘤内异质性、肿瘤治疗反应、肿瘤转移和肿瘤转移的理解方面取得巨大进展,以及肿瘤亚群相互作用在驱动肿瘤发生和识别潜在有用生物标志物中的作用。
原文链接请参阅:https://clinicalepigeneticsjournal.biomedcentral.com/articles/10.1186/s13148-021-01120-7
Active Motif质谱解决方案
如果这项质谱研究引起了您的兴趣,可查看Active Motif提供的Mod Spec®服务。Mod Spec®可以测量80多种不同组蛋白修饰的相对丰度,用于探究疾病或治疗过程中发生的表观遗传变化。同时,最近开发的RIME(内源性蛋白质的快速免疫沉淀质谱)技术可以帮助识别转录共因子和染色质相关蛋白。Mod Spec®检测得到的大量组蛋白修饰信息可帮助研究人员确定预期的表观变化,而且还能够帮助研究人员发现意想不到的变化,这可能会为研究提供更多有用且有趣的信息。
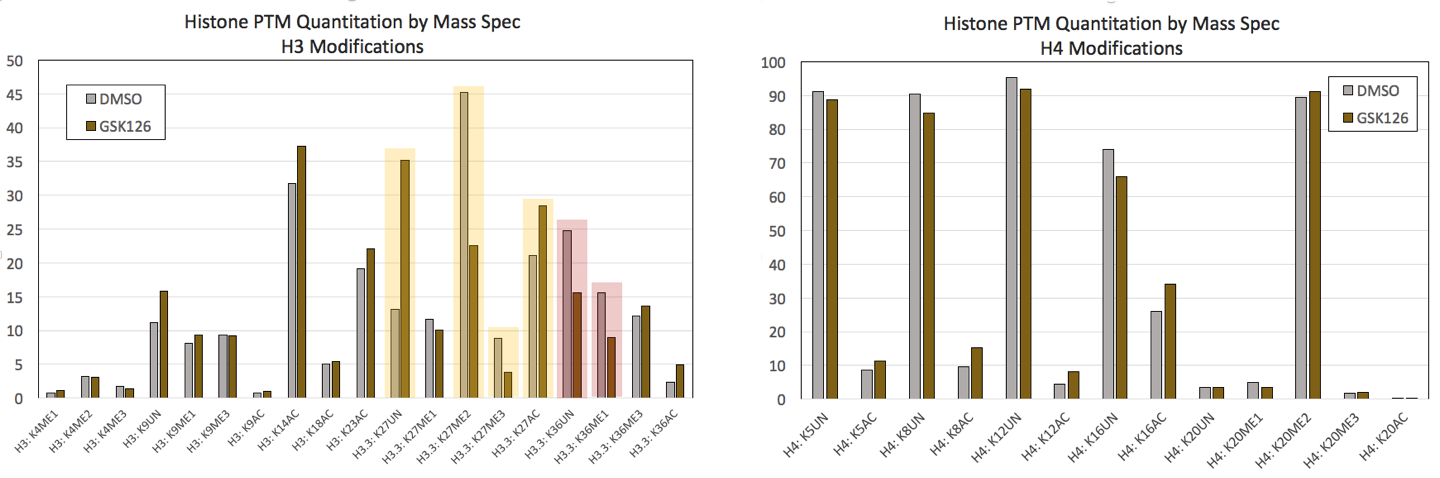
来自对照HeLa细胞和用0.5 μM GSK-126处理7天的HeLa细胞的Mod Spec®数据。(点击图片可放大)
GSK126是一种阻断EZH2甲基转移酶活性的抑制剂,导致H3K27me2和H3K27me3的整体降低。A) 从Active Motif的Mod Spec®分析中可以选择H3修饰,确认H3K27甲基化显著降低,同时乙酰化和未修饰的H3K27(以黄色突出显示)增加。还检测到H3K36的变化(以红色突出显示)。B) 选择的H4修饰对GSK126处理的反应没有显示出显著的变化。
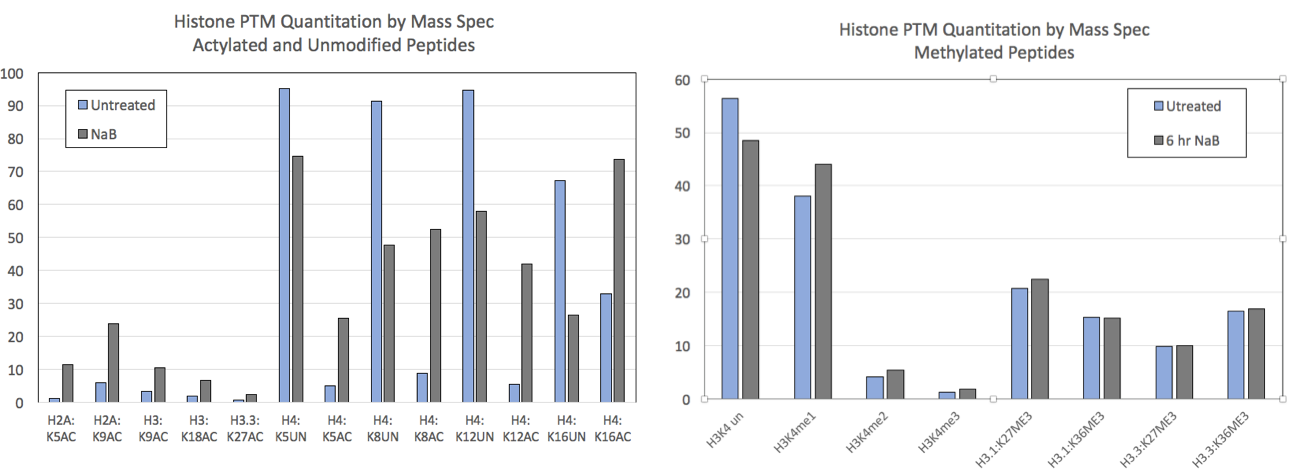
Mod Spec®数据来自对照HEK293细胞和用5 mM NaB处理6小时的HEK293细胞。
NaB是一种通用的HDAC抑制剂,NaB处理可提高组蛋白乙酰化水平。左图 显示了选择乙酰化和未修饰组蛋白位点的Mod Spec®数据。处理后所有位点的乙酰化都增加。未经修饰的肽在某些位置有信号,这些位置并在NaB处理后显示相应的减少。B) 不同组蛋白残基的甲基化不受NaB处理的影响。
参考文献
[1] Mara, Colzani, Roberta, et al. Quantitative Chemical Proteomics Identifies Novel Targets of the Anti-cancer Multi-kinase Inhibitor E-3810[J]. Molecular & Cellular Proteomics, 2014, 13(6):1495-509.
[2] Roberta, Noberini, Rémi Longuespée, Cristina Richichi, et al. PAT-H-MS coupled with laser microdissection to study histone post-translational modifications in selected cell populations from pathology samples[J]. Clin Epigenet 9, 69 (2017).
[3] Roberta, Noberini, Camilla, et al. Enrichment of histones from patient samples for mass spectrometry-based analysis of post-translational modifications[J]. Methods (San Diego, Calif.), 2019.
[4] Restellini C, Cuomo A, Lupia M, et al. Alternative digestion approaches improve histone modification mapping by mass spectrometry in clinical samples[J]. Proteomics. Clinical applications, 2018.
[5] Noberini R, Restellini C, Savoia E O, et al. Profiling of Epigenetic Features in Clinical Samples Reveals Novel Widespread Changes in Cancer[J]. Cancers, 2019, 11(5).
[6] Fowler, C.B., O’Leary, T.J., Mason, J.T. (2011). Protein Mass Spectrometry Applications on FFPE Tissue Sections. In: Al-Mulla, F. (eds) Formalin-Fixed Paraffin-Embedded Tissues. Methods in Molecular Biology, vol 724. Humana Press.