上海艾跃(Active Motif)生物科技有限公司品牌商
10 年
手机商铺
- NaN
- 0.3999999999999999
- 0.3999999999999999
- 2.4
- 2.4
上海艾跃(Active Motif)生物科技有限公司
入驻年限:10 年
- 联系人:
Active Motif
- 所在地区:
上海 闵行区
- 业务范围:
试剂、实验室仪器 / 设备、技术服务、抗体
- 经营模式:
生产厂商 经销商
推荐产品
公司新闻/正文
Active Motif ChIP 实验技术 : ChIP-Seq 样本间如何做定量比较?
5148 人阅读发布时间:2018-10-30 15:30
引言
Active Motif 与Constellation制药合作开发了一种新的用于ChIP-Seq的标准化数据校正技术。将这种技术应用于检测药物处理前后组蛋白修饰的变化,成功地检测出人源细胞全基因组水平上H3K27me3的变化。这种全新的方法可以帮助研究者对经过不同处理的样本的ChIP-Seq数据做比较,揭示用传统方法无法检测到的变化。
文章2016年11月22日发表于PLOS one杂志。

原文链接:http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0166438
>>> 标准ChIP-Seq方法检测不到EZH2抑制剂处理前后H3K27me3水平的变化
EZH2是组蛋白H3K27特异性的甲基转移酶,在转录抑制上发挥着重要的作用。基因组学和转录组学数据都将EZH2识别为很多人类恶性肿瘤的候选靶点。如前列腺癌,乳腺癌以及血液恶性肿瘤。在扩散的B细胞淋巴瘤,滤泡性淋巴瘤以及黑色素瘤中也发现了复发的体细胞EZH2突变。
目前,许多EZH2的小分子抑制剂被应用于临床研究,被认为是很有潜力的肿瘤治疗药物。为了更好的了解EZH2的抑制作用对肿瘤细胞的影响,检测抑制剂引起的全基因组范围H3K27me3变化是非常重要的。
然而,使用传统的ChIP-Seq 步骤和分析方法来检测全基因组H3k27me3水平的变化被证实是非常困难的。
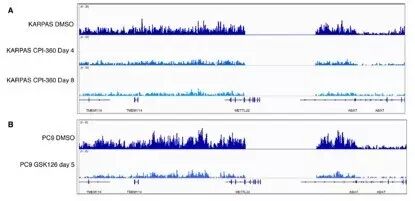
研究人员使用B细胞淋巴瘤细胞KARPAS-422和肺癌细胞PC9,分别用不同的EZH2抑制剂CPI360和GSK126处理之后用不同方法检测总体H3K27me3的变化。
WesternBlot检测结果显示两种细胞在加入抑制剂之后H3K27me3水平出现了显著的降低。
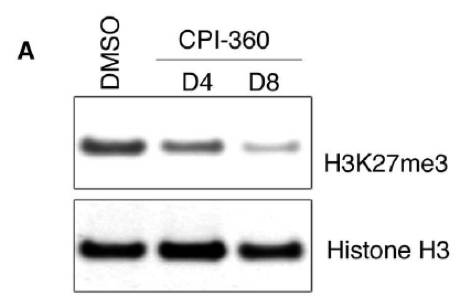
| A. KARPAS-422细胞使用1.5uM CPI-360分别处理4天和8天 |
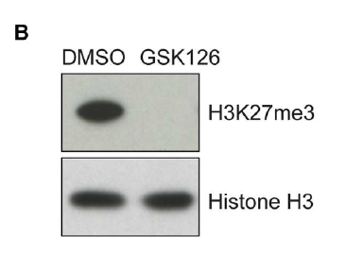
| B. PC9细胞使用1uM GSK126处理5天 |
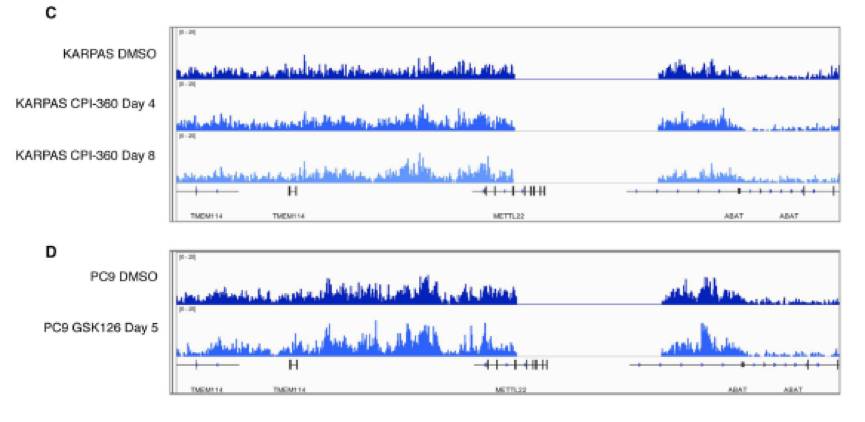
然而,当使用ChIP-Seq方法来想进一步检测加入抑制剂后H3K27me3在全基因组范围的分布变化的时候,却发现加入抑制剂样本组和加入DMSO样本之间没有差别。
>>> 为什么ChIP-Seq检测不到变化?



根据之前的报道,抑制剂处理之后,一些特异性位点的修饰水平发生了变化。通过qPCR的方式对该位点进行检测,两种抑制剂处理之后都检测到了H3K27me3的降低。
然而对建库之后的样本进行qPCR检测却检测不到变化。这说明ChIP-Seq检测不到变化很有可能是建库过程引起。
文库构建需要PCR扩增,导致不同样本之间的真实差异发生变化。抑制剂处理的样本免疫沉淀下来的DNA量是少于未处理样本的,但是建库之后,两组样本的DNA量是相同的。
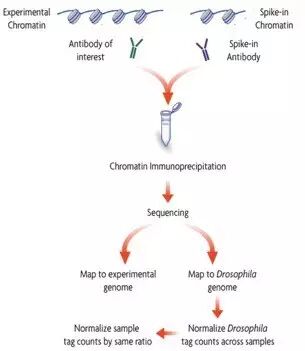
>>> Active Motif Spike-in技术
一个看起来很直接的解决问题的方法是减少建库过程中的PCR循环数。
然而事实上,即使建库之后的DNA保留了建库之前沉淀下来的DNA量的差别,这种差别也会在cluster generation时丢失。
为了保证同一泳道不同样本之间数据量相同,每个样本的上样量是一样的。
为了解决这一问题,Active Motif 开发了果蝇染色质Spike-In技术,通过加入对照“保留”样本之间的差别。
标准的ChIP反应是使用样本染色质和抗体建立的。在“Spike-in”校正体系中,黑腹果蝇(Drosophila melanogaster)染色质在免疫反应之前加入到样本染色质中混合。
免疫沉淀时不仅加入目标蛋白特异性抗体,同时也加入特异性的果蝇组蛋白变体H2Av抗体,这样目标蛋白抗体沉淀目标蛋白结合的DNA,果蝇组蛋白变体H2Av抗体沉淀出部分果蝇染色质。
沉淀出的所有DNA用于建库和测序。
测序之后,序列标签被分别比对到样本(如:人,小鼠等)基因组和果蝇基因组。因为不同样本之间加入的起始果蝇染色质是一样多的,理论上沉淀下来的DNA量也应该是一样的,这样不同样本之间的果蝇序列标签数就可以作为标准,用于校正样本之间的标签序列数。
使用这一方法成功检测到了EZH2抑制剂处理后全基因组H3K27me3水平的变化。
Spike-in 技术已经应用于Active Motif 提供的ChIP-Seq技术服务。基于广大用户的需求,近期Active motif 也推出了Spike-in 产品。
>> 往期ChIP实验相关内容:
【Active Motif 实验技术】ChIP 实验染色质超声破碎指南(上篇)
【Active Motif 实验技术】: ChIP 实验染色质超声破碎指南(下篇)
【Active Motif 实验技术】如何判断ChIP是否成功?
【Active Motif 实验技术】如何才能做好 ChIP 实验?
| 作者招募 Active Motif 欢迎表观遗传领域小伙伴们前来投稿,分享伙伴们的研究进展、学术见解、实验方法、实验趣事、研究路上经历故事... ... ◆ 投稿请联系:wzhao@activemotif.com◆ 微信可添加_表观小秘书:18521362870 期待您的加入~~ |
扫一扫下方微信二维码

Tel:021-20926090
Email:Techchina@activemotif.com